Diversity指数学习笔记
Diversity指数学习笔记
简述
假设:我们手中有肿瘤样本在某药物治疗前和治疗后的配对TCR数据,想比较药物对患者治疗前后的CDR3多样性的影响。我们需要考虑的的问题有以下两个:
- 衡量治疗前和治疗后,对于相同样本,CDR3序列多样性(CDR3 repertoire)变化程度如何?是有很大差异,还是该药物没有对CDR3多样性造成影响?
Alpha diversity(别名within sample diversity) - 不同样本之间CDR3是相似,还是差距很大?
Beta diversity
Alpha diversity

如上图:两个花园里花的种类一样丰富吗?
对于Alpha多样性,常用的指标/指数是Shannon, Inverse Simpson, Simpson, Gini, Observed and Chao1。
这些指标一般由两个部分的度量组成:
用于衡量样本内有多少种unique CDR3数量的指标:丰富度(richness)。(每种CDR3是否存在,如下图,用1或0表示)
用于衡量样本内每种CDR3分布频率的指标:均匀度(evenness)。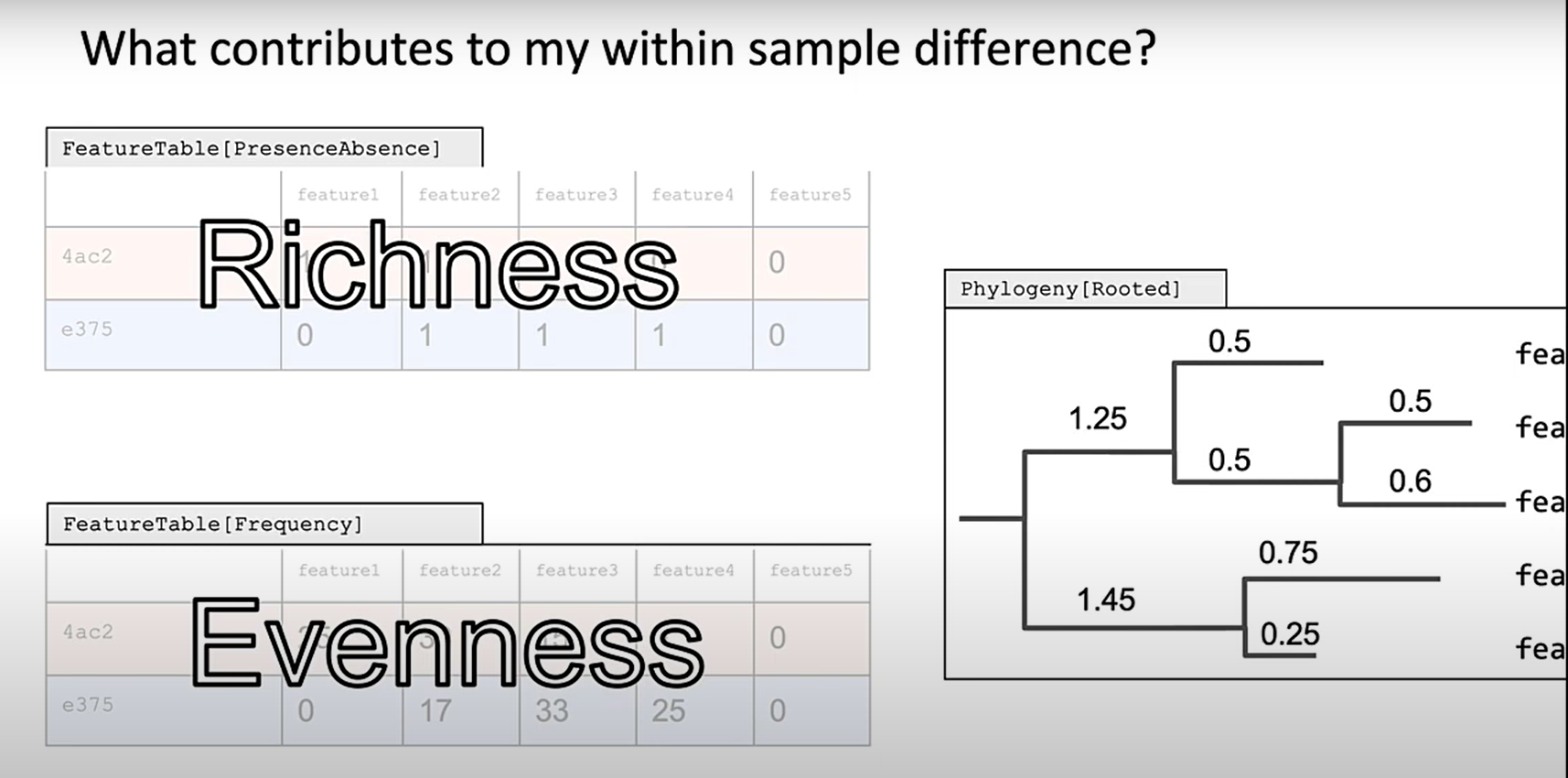
观察下面的两个树群:树群1和树群2的richness相同,但evenness不同。树群1的树种类分布要更加均衡,而树群2的diversity要更高。
高Evenness(范围为 0 到 1)意味着所有元素几乎均匀分布,而低均匀度则表明向某一群体偏斜。从下图可以看出,树群2的第三种树占据了70%的比例,因此树群2的evenness要更低,树群1的evenness更高。
Shannon entropy
Shannon entropy 此指标同时考虑 richness和evenness, 但是更重视evenness。该指数越高,说明样本中克隆的diversity越高。
根据下图展示的公式,我们需要如下信息来计算香农熵:
- CDR3的总数:n
- 每种CDR3的频率:pi (一般我们是通过CDR3 counts来计算的)
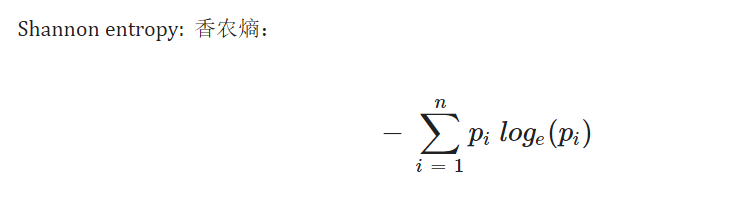
按照公式对上面的树木分布计算香农熵,Community 1的值是1.39,Community 2的值是1.06。(由于树群1和树群2的richness相同,但树群1的evenness更高,所以树群1的shannon entropy要更高。)
在实现上可以使用immunarch的entropy函数,为每个sample计算对应的shannon entropy
源码见:
https://github.com/immunomind/immunarch/blob/HEAD/R/info_theory.R
1 | |
Simpson’s Diversity Index
Simpson index同时考虑Richness和Evenness
公式如下: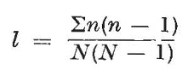
我们通常不直接用Simpson index(D),它反映的是在同一个样本中随机的抽取2个个体,这两个个体来自同一个类的概率(以TCR为例,辛普森指数是从样本中随机选择的任意两个tcr具有不同克隆型的概率。)。故D值越大,多样性越低。这与直觉和逻辑不符。
为了解决这个问题,通常会用以下两种形式来表示:
(1)1-D,即Gini-Simpson Diversity index
Gini-Simpson多样性指数表示在克隆群中随机选取两个克隆序列,两者属于不同种类克隆的概率。Gini-Simpson值越高,代表克隆的多样性越高。公式中,ni为第i个特异性克隆类型的氨基酸序列的总数,N为样本中总的序列数。(使用时有些地方也会将Gini-Simpson简化成Simpson,实际上这里是指Gini-Simpson,可根据公式区分,比如此处就直接缩写为simpson了:示例)
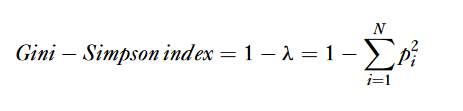
可使用immunarch直接计算
div_div <- repDiversity(immdata$data, “gini.simp”)
数值接近0 表示没有多样性(即高度寡克隆),数值接近1就是diversity接近无穷(polyclonal repertoire with equivalent representation of each clone)
(2)1/D,即Inverse-Simpson index
可使用immunarch直接计算
div_div <- repDiversity(immdata$data, “inv.simp”)
逆辛普森多样性指数是辛普森指数的倒数,逆辛普森指数越大,表示克隆的多样性越高。侧重于反映高频克隆的多样性。
Hill numbers
hill number是一个比较广义的度量,公式如下: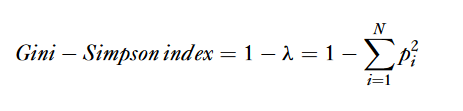
如下图,可以看到横坐标是Q value。hill number量化了diversity。丰度分布的重要性随着希尔阶数(Q,也就是公式中的alpha,即阶数)的增加而增加。对于 Q=0,希尔数是richness;对于 Q =1,它其实就是Shannon entropy,就可以解释为常见或较丰富CDR3序列的有效数量 ;对于 q=2,它是inverse Simpson index,可以解释为优势或高度丰富CDR3的有效数量。
因此,hill number就是比较在同样的diversity下,CDR3序列的数量在哪个样本中更高。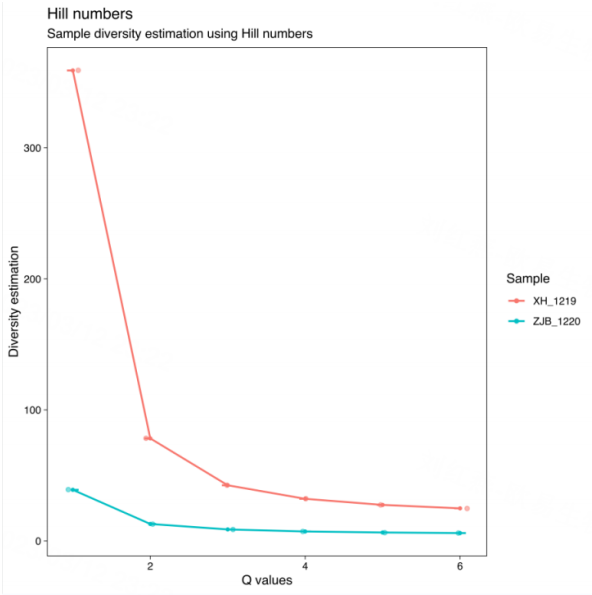
Diversity evenness 50 (DE50)
此指标仅考虑Evenness。
DE50值越高,代表各克隆分布比较均匀,DE50值越低,克隆性越高,代表有一些特异性的克隆发生了扩增(这种特异性克隆扩增的情况可以形容为“更具寡克隆性”,oligoclonal)。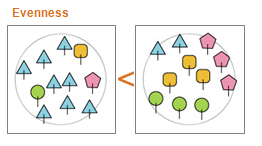
公式的计算方法为:将克隆序列按照频率从高到低排序,从最高开始累加,达到频率总和为50%的这些序列的克隆种类占总克隆种类的比例。(Diversity Evenness 50 (DE50) was calculated as the ratio of how many clonotypes amongst the most frequent were necessary to account for 50% of the total read counts divided by the total number of read counts present.)
此指标在immunarch中也叫D50,用repDiversity函数计算:
d50 is a recently developed immune diversity estimate. It calculates the minimum number of distinct clonotypes amounting to greater than or equal to 50 percent of a total of sequencing reads obtained following amplification and sequencing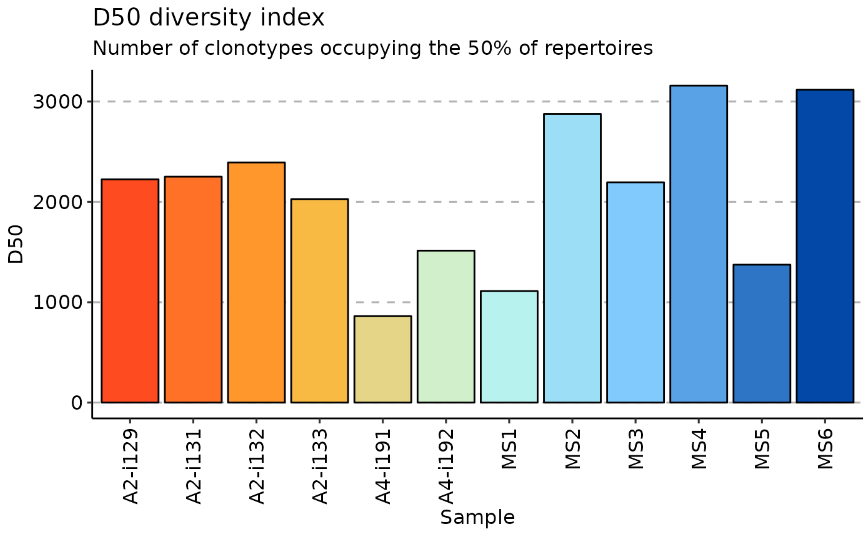
Pielou’s index
与D50一样,此指标仅考虑Evenness。
Pielou 指数是衡量物种在群落中分布均匀程度的一种方法。 Pielou 指数值定义在 0 到 1 之间。1 代表具有完美均匀度的群落,随着物种的相对丰度偏离均匀度,该指数会降至零。
In parallel to DE50, clonal evenness of a repertoire can be calculated using Pielou’s index, which is itself derived from the ratio between the Shannon entropy and the maximization of the diversity distribution of species within a sample
Pielou 指数是基于 Shannon entropy计算 ,使用 Shannon entropy 除以样本内的总克隆型的数量(N),计算公式如下: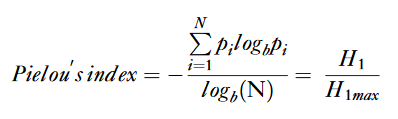
与Pielous指数相关的另一个概念是“Clonality”,克隆性:
计算Clonality可以使用1-Pielou 指数。
该数值越接近0,evenness越高;数值越接近1,即存在优势克隆(clonal dominance),少数克隆出现在较高频率。
计算公式如下: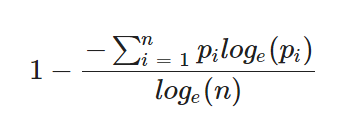
典型文献是在对接受PD-1免疫治疗药物治疗的转移性黑色素瘤患者的外周和肿瘤 T 细胞克隆性时,强调了以 1-Pielou 指数为代表的克隆扩张与临床反应之间的关联。实验结果显示,在药物治疗后出现更高的克隆性的患者,具有更长的生存时间。
Gini coefficient
此指标仅考虑Evenness。
注意gini coefficient(基尼系数) 与 Gini-Simpson不是同样的概念。
与上面提到的Pielou 指数类似,gini coefficient也可以用作clonality的度量
基尼系数的范围从 0(CDR3 repertoire的最大多样性,即每个序列的丰度相等) 到 1(即极端不平等, 出现对单个CDR3序列的高克隆性)
Chao1
此指标仅考虑Richness。
用来反应 clonotypes 的丰度,对稀有的物种很敏感。
Chao1 指数越高,clonotypes Richness越高,多样性越高。
受到Sample size的影响较大,在high diverse的群体中不准确,因此一般不单独使用,都是和其余指标联合使用。
多指数评估文献1
典型文献
Beta diversity

如上图:两个花园里是否有相同的花?
β -多样性:衡量不同群体样本之间的差异,以确定整体群落组成和结构是否存在差异。
名词解释
Jensen-Shannon divergence
白板推导教程参考:
https://www.youtube.com/watch?v=bqjZK9tkWdk
除了对每个样本进行的diversity分析之外,TCR/BCR 测序数据还需要进行相似性分析,以便比较 T-cell repertoires之间的重叠。JSD指数就是一个反应相似性的指数,**JSD越低,repertoire stability越高,也就是说两个CDR3氨基酸频率分布的差异越小(也可以说V和J段的分布更相似。)**
示例一: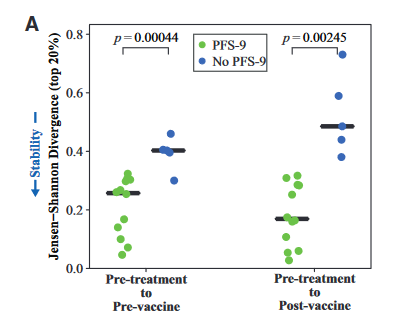
上面的例图展示了 PFS-9患者(药物治疗后疗效更好的患者组) 和 非PFS-9患者(药物治疗疗效较差的患者组) 疫苗注射前(左) 或 疫苗注射后(右) 与基线(Pre-treatment)之间的TCRb CDR3序列的JSD值。如左侧箭头所示,低JSD值表示repertoire stability。黑线为中位数。p值来自双尾Student ‘s t检验。
本图使用philentropy包实现,此包可以实现两组比较的效果(由于JSD的对称特性,比较组不分先后)
示例二: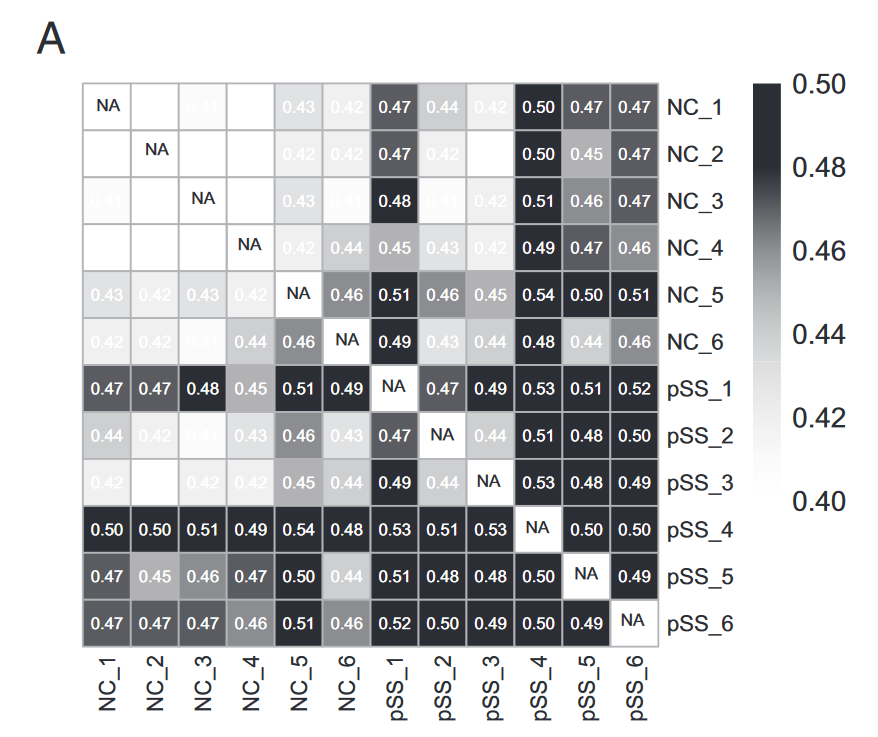
本图使用immunarch实现(immunarch对所有输入的样本进行成对检验,出具热图):
https://immunarch.com/articles/web_only/v5_gene_usage.html?q=divergence#gene-usage-analysis